Équipe Philippe MARIN
Neuroprotéomique et signalisation des pathologies cérébrales
Projet Régulations et signalisation du récepteur mGlu2 du glutamate, cible d’une nouvelle génération d’antipsychotiques
RESPONSABLE

Personnel IGF impliqué
Philippe MARIN
DRCE CNRS
Candice DISDIER
Doctorante Inserm
Sonya GALANT
IE CNRS
Notre projet actuel vise à décrypter in vivo la signalisation du récepteur métabotropique du glutamate mGlu2, cible d’une nouvelle classe d’antipsychotiques actuellement en test clinique contre la schizophrénie.
Il utilise la spectrométrie de masse haute résolution et des nanocorps (minuscules anticorps de lama) afin de caractériser in vivo le profil de phosphorylation du récepteur, ses protéines associées et les voies de signalisation engagées lors de son activation.
In fine, ce projet permettra de comprendre comment agissent les antipsychotiques ciblant le récepteur mGlu2 particulièrement sur les symptômes résistants aux antipsychotiques actuels.
Parmi les protéines interagissant avec le récepteur mGlu2 caractérisées dans le cortex préfrontal, nous avons identifié le récepteur TrkB de neurotrophines. Les deux récepteurs renforcent réciproquement leurs activités de signalisation. L’activité du récepteur TrkB s’est montrée importante pour la réponse comportementale d’un modèle murin aux ligands mGlu2 en particulier pour les perturbations relatives aux symptômes négatifs et cognitifs de la schizophrénie.
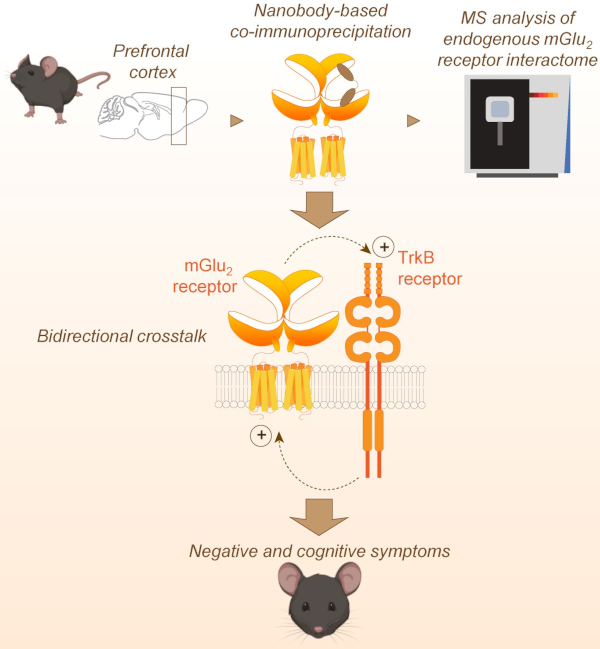
Procédure expérimentale utilisée pour caractériser l’interactome du récepteur mGlu2 dans le cortex préfrontal de souris
Principales publications
• Philibert C., et al. (2024) Sciences Advances. 10(4):eadg1679.
• Schlotter F., et al. (2023) Nucleic Acids Res. 51(7):3357-3374.
• Dos Santos Morais R., et al. (2022) J Mol Biol. 434(19):167760.
• Klimešová K., et al. (2021) Nat Commun. 12(1):3646.
• Abel Y., et al. (2021) Nucleic Acids Res. 49(2):1094-1113.
• Murat S., et al. (2019) Mol. Psychiatry, 24(11):1610-1626.
• Henri J., et al. (2018) Structure. 26(9):1196-1209.e8.
• Cassier E., et al. (2017) Elife, 6:e23777.
• Karaki S., et al. (2014) Mol Cell Proteomics. 13(5):1273-85.
Financements
• Canceropole GSO project SykOnGPCRs 2023-2024. Coordinator
• PRC ANR-22-CE14 MOSDER 2022-2026. Partner
• PRC ANR-21-CE44-0006-02 InteracT 2021-2025. Partner
• PRC ANR-20-CE44-0006 SchizoGlu 2021-2024. Coordinator
• Isite Montpellier UniverSité d’Excellence project NanoSchizo 2018-2022. co-PI
• PRC ANR-10-BLAN-1619 GPCRnet 2011- 2015. Partner
• PRC ANR-2010-BLAN-1601 PhosphoCav 2010-2014. Partner
Collaborations
• Philippe RONDARD / Jean-Philippe PIN (Montpellier)
• Emmanuel BOURINET (Montpellier)
• Céline VERHEGGEN / Edouard BERTRAND (Montpellier)
• Séverine MASSENET (Nancy)
• Sandra LECAT / Frederic SIMONIN (Strasbourg)
• Frédéric JEAN-ALPHONSE / Eric REITER (Tours)
• Evi KOSTENIS (Bonn)
• Ferdinando NICOLETTI (Rome)
• Jiang-Feng LIU (Wuhan)
Alumni
• Thomas Le Cler (Master 1 2023)
• Clémentine Philibert (PhD student 2018-2022)
• Jeanne Espinosa (Master 1 2022)
• Candice Disdier (Master 2 2021)
• Elisabeth Cassier (IE 2009-2017)
• Mathilde Bigot (Master 1 2016)
• Samy Murat (PhD student 2014-2018, Master 1, 2013)
• Samah Karaki (PhD student 2009-2011)
• Jonathan Chapron (Master 1 2015)
• Ramzi EL Fegahli (IR CDD INSERM 2011-2013)
• Madeleine Sugano (IE CDD 2011-2012)
• Alexis Couthier (Master 2 2012)
• Guillaume Blanc (IE CDD 2011)
• Simon Perrin (Master 2 2011)
• Simon Bruno (Master 1 2010)


