Équipe Laurent JOURNOT
Génomique fonctionnelle des gènes soumis à empreinte parentale
Projet Méthodes statistiques pour les données de génomique
RESPONSABLES

Christelle REYNES
MCU UM

Laurent JOURNOT
DR CNRS
Personnel IGF impliqué
Robert SABATIER
PU UM
Anne LE DIGARCHER
AI CNRS
Tristan BOUSCHET
CR INSERM
Dans le cas des gènes soumis à empreinte génomique parentale, l’identification des biais alléliques dans les données transcriptomiques n’est pas un problème trivial et nécessite le développement de méthodes spécifiques. Ces méthodes sont difficiles à paramétrer, ce qui a conduit à une controverse sur le nombre de gènes soumis à empreinte dans les génomes de mammifères. De même, la définition de modules fonctionnels dans les réseaux de gènes co-exprimés pose des défis spécifiques qui peuvent bénéficier de la mise en œuvre de techniques basées sur l’apprentissage automatique.
Nous avons développé ISoLDE (Integrative Statistics of alleLe Dependent Expression), une méthode statistique non paramétrique qui déduit directement le déséquilibre allélique des données RNA-seq. ISoLDE apprend à partir des données la distribution d’une statistique de test spécialement conçue et qualifie l’expression des gènes de biaisée, bi-allélique ou indéterminée. ISoLDE est disponible sous la forme d’un paquet Bioconductor.
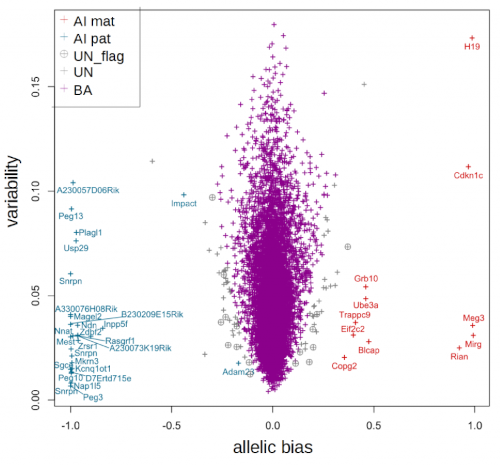
Graphe produit par la version de rééchantillonnage d’ISoLDE. Pour chaque gène, la variabilité (valeur du dénominateur de la statistique Sg) a été tracée en fonction du biais allélique (valeur du numérateur de la statistique Sg). Les croix violettes correspondent aux gènes exprimés de manière bi-allélique (« BA »). Les croix rouges et bleues correspondent à des gènes appelés déséquilibrés maternellement et paternellement (« AI mat » et « AI pat », respectivement). Les croix grises correspondent à des gènes indéterminés (‘UN’). Les croix grises cerclées correspondent aux gènes indéterminés présentant cependant un biais dont le sens est constant ou dont la significativité est limite (« UN_flag »).
Nous avons également développé TopoFun, une méthode d’apprentissage automatique pour identifier les modules fonctionnels dans les réseaux de co-expression génique et compléter les annotations Gene Ontology.
Une annotation fonctionnelle complète et précise des gènes est essentielle pour les approches systémiques. La génétique a produit une quantité substantielle de données sur les fonctions des gènes ; cependant, une grande partie des gènes sont encore mal annotés, même dans les organismes modèles. Une approche possible pour compléter les annotations existantes consiste à analyser la coexpression des gènes, les gènes fonctionnellement liés ayant tendance à être co-exprimés.
Les données relatives à la coexpression des gènes sont représentées sous la forme de graphes à grande dimension dans lesquels les nœuds représentent les gènes et les arêtes leur co-expression. TopoFun est une méthode d’apprentissage automatique qui combine des informations topologiques et fonctionnelles sur les modules de coexpression. Nous avons d’abord sélectionné des descripteurs topologiques des modules de coexpression génique qui permettent de distinguer les modules constitués de gènes fonctionnellement liés des modules constitués de gènes sélectionnés de manière aléatoire. En utilisant les descripteurs topologiques sélectionnés, nous avons construit une base de données de modules fonctionnels et aléatoires et effectué une analyse discriminante linéaire pour prédire le type d’un module. À partir d’un processus biologique Gene Ontology (GO-BP) donné, nous avons utilisé un algorithme génétique pour trouver des gènes dont la coexpression avec la plus grande clique du GO-BP suggère qu’ils peuvent être fonctionnellement liés.
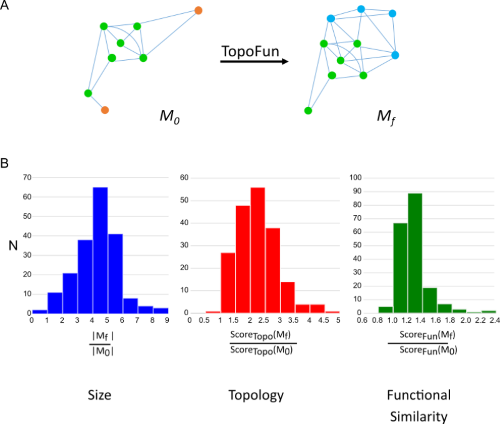
La méthode d’apprentissage automatique TopoFun. A. En partant d’un module de gènes co-exprimés M0, TopoFun a éliminé les gènes qui n’étaient que marginalement connectés à la plus grande clique du module et a ajouté de nouveaux gènes qui étaient à la fois fortement connectés à ceux de la plus grande clique et fonctionnellement similaires, produisant ainsi le module Mf. B. Distribution du ratio de taille, du ratio ScoreTopo et du ratio ScoreFun. Nous avons exécuté TopoFun sur 193 GO-BP comprenant 50 à 100 gènes. Pour chaque M0 (=GO-BP) et Mf (= module ‘optimal’), nous avons déterminé le nombre de gènes, le ScoreTopo, et le ScoreFun, et tracé la distribution des ratios de ces variables pour Mf par rapport à M0. Les figures montrent que les rapports sont le plus souvent >1, ce qui indique que TopoFun augmente la taille des modules et améliore la topologie et la similarité fonctionnelle.
Principales publications
• Janbain A et al. (2021) NAR Genom Bioinform, 3, lqab103.
• Reynès C et al. (2020) Bioinformatics, 36, 504.
Alumni
• Ali Janbain (PhD student, 2014-2019)


