Équipe Laurent JOURNOT
Génomique fonctionnelle des gènes soumis à empreinte parentale
Projet Le réseau des gènes à empreinte
RESPONSABLE

Personnel IGF impliqué
Anne LE DIGARCHER
AI CNRS
Tristan BOUSCHET
CR INSERM
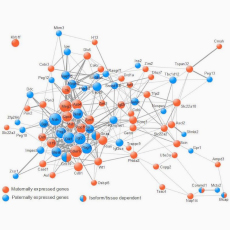
Notre équipe se concentre sur les fonctions des gènes imprimés et vise à identifier les processus biologiques qu’ils contrôlent. Ceci est principalement motivé par les nombreux syndromes humains qui résultent de défauts d’empreinte ou d’altérations cytogénétiques des loci imprimés (Mackay & Temple, Eur J Med Genet, 2017). Les phénotypes de ces syndromes sont complexes et diversifiés (Peters, Nat Rev Genet, 2014) ; cela suggère que les gènes imprimés ont des fonctions diverses et peuvent ne pas être fonctionnellement liés. D’autre part, les mutants de gènes à empreinte présentent des caractéristiques communes, à savoir des défauts de croissance embryonnaire et un équilibre énergétique déréglé (Plasschaert & Bartolomei, Development, 2014).
Une interprétation possible est que les gènes à empreinte remplissent différentes fonctions dans différents types de cellules, comme le montre la figure 1. Selon ce point de vue, chaque gène imprimé aurait une fonction clé dans le(s) tissu(s) le(s) plus affecté(s) par le mutant correspondant. Le contrôle de la croissance de l’organisme serait assuré par un ensemble de processus apparemment divers, comme le suggère l’absence de termes communs de la Gene Ontology associées aux gènes à empreinte.
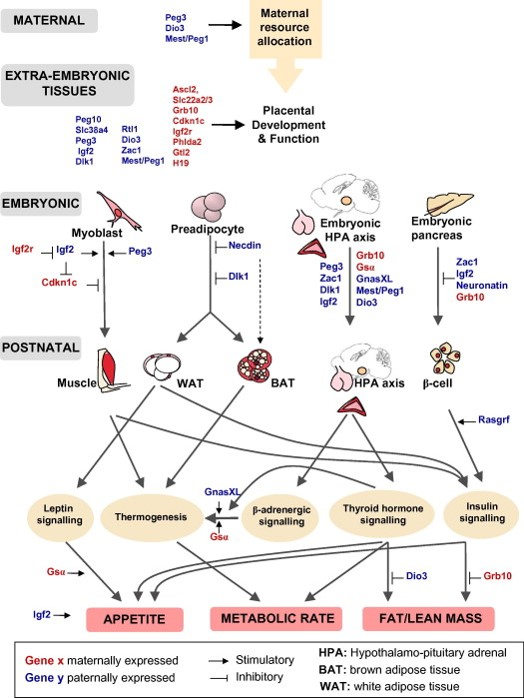
Les gènes imprimés sont essentiels pour le contrôle de l’équilibre énergétique. Le dosage correct de l’expression des gènes imprimés est vital pour l’homéostasie énergétique au cours de la vie prénatale et postnatale. Les gènes imprimés jouent un rôle essentiel dans le contrôle de l’approvisionnement en nutriments du fœtus par le biais d’effets sur le métabolisme maternel et la répartition de l’énergie, ainsi que sur le développement et la fonction du placenta. Il existe également des preuves que les gènes imprimés agissent de manière coordonnée chez le fœtus pour réguler la croissance, modifiant ainsi la demande du fœtus en ressources maternelles. Les gènes imprimés jouent un rôle clé dans le développement des organes métaboliques et modulent les principales voies métaboliques chez l’adulte. Tiré de Radford EJ, Ferrón SR, Ferguson-Smith AC (2011) Genomic imprinting as an adaptive model of developmental plasticity (L’empreinte génomique en tant que modèle adaptatif de la plasticité développementale). FEBS Lett. 585:2059-66
Nos travaux (Al Adhami et al., Genome Res., 2015) soutiennent un mécanisme alternatif dans lequel les gènes à empreinte contrôlent de manière coopérative un processus biologique unique, qui a un impact sur la fonction de divers organes impliqués dans le contrôle de la croissance de l’organisme. En utilisant des données issues de méta-analyses de puces à ADN, nous avons montré que les gènes imprimés sont fréquemment co-exprimés (Figure 2).
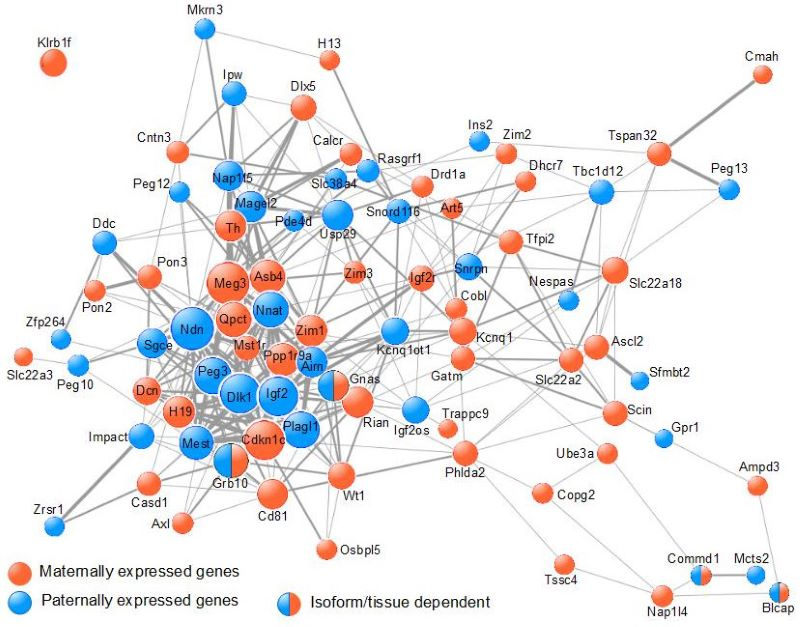
Les gènes imprimés sont fréquemment co-exprimés. La méta-analyse COXPRESdb des données de microréseaux a été recherchée pour la co-expression parmi les IG murins. Les liens de co-expression qui en résultent sont représentés à l’aide de Cytoscape. La taille des nœuds est proportionnelle à leur degré. La largeur des arêtes représente le rang mutuel entre deux nœuds donnés.
Pour démontrer que la co-expression des gènes imprimés observée est significative, nous avons comparé la topologie de ce réseau à celle obtenue avec des ensembles de gènes connus pour être fonctionnellement liés, c’est-à-dire des gènes appartenant au même « processus biologique » tel que défini par Gene Ontology, ou à des ensembles de gènes sélectionnés de manière aléatoire (Figure 3).
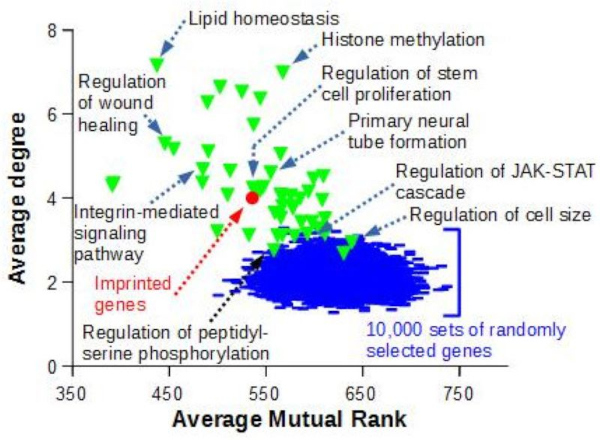
Le degré moyen et le rang mutuel moyen ont été calculés pour le réseau de 85 gènes à empreinte présenté dans la figure 2 (cercle rouge). Des réseaux aléatoires (lignes bleues) ont été générés en tirant au hasard 10 000 ensembles de 85 GeneID avec des données dans COXPRESdb, et en récupérant les liens de co-expression comme pour les 85 gènes à empreinte murine. Un travail similaire a été effectué avec 50 ensembles de 85 gènes compris dans GO Biological Processes (triangles verts) et avec des données dans COXPRESdb.
Nous avons également montré que les gènes imprimés sont régulés de manière coordonnée lors de la transition de la prolifération à la quiescence et à la différenciation pendant le retrait du cycle cellulaire des fibroblastes, l’adipogenèse in vitro (figure 4) et la régénération musculaire in vivo.

Carte thermique des niveaux d’expression des gènes imprimés au cours de la différenciation adipogénique de 3T3-L1. Les préadipocytes 3T3-L1 ont été cultivés de manière exponentielle (P, prolifération) jusqu’à ce qu’ils atteignent la confluence (Q, quiescence) après inhibition du contact. Quarante-huit heures plus tard, ils ont été induits à se différencier suite à l’ajout d’IDX (Insuline, Dexaméthasone, IBMX), ont repris la prolifération pendant la phase d’expansion clonale (CE, expansion clonale), et ont finalement quitté le cycle cellulaire et se sont différenciés (D, différenciation). Les niveaux d’expression des gènes imprimés indiqués ont été contrôlés par PCR en temps réel et exprimés en % des niveaux d’expression maximaux pour ce gène. Cebpd, Pparg, Lpl, Cebpa, Plin1, Adipoq, Slc2a4 et Lep sont des marqueurs de la différenciation adipogénique précoce et tardive.
La régulation des gènes imprimés n’est pas liée à l’altération de la méthylation de l’ADN ou à la perturbation de l’expression mono-allélique dépendante du parent d’origine. La surexpression et le knockdown de l’expression des gènes imprimés modifient la sensibilité des préadipocytes à l’inhibition du contact et à la différenciation adipogénique (figure 5).
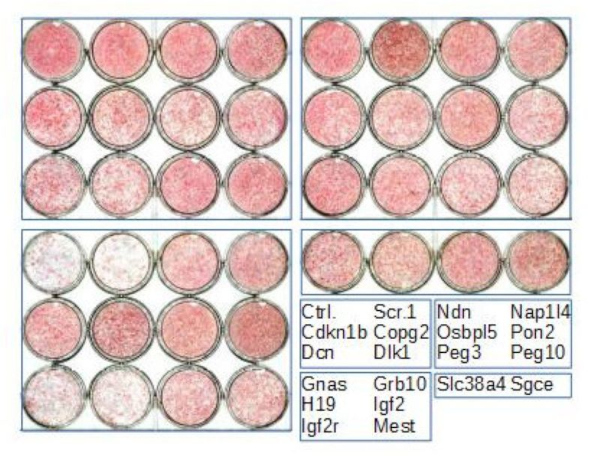
Effet de la régulation à la baisse des gènes imprimés sur la différenciation adipogénique des préadipocytes 3T3-L1. Coloration représentative à l’Oil Red O (ORO, un colorant lipidique) de cellules 3T3-L1 non transfectées (Ctrl.) ou transfectées avec un siRNA contrôle (Scr.1) ou avec des siRNA ciblant les gènes indiqués, placées en double à confluence, incubées avec IDX 3 jours après la transfection, et fixées 12 jours plus tard.
Des expériences in silico et in cellulo ont montré que le réseau de gènes imprimés comprend des gènes non imprimés exprimés de manière bi-allélique, qui contrôlent la composition de la matrice extracellulaire, l’adhésion cellulaire, la jonction cellulaire et la signalisation activée par la matrice extracellulaire et le facteur de croissance. Ces observations montrent que les gènes à empreinte partagent un processus biologique commun qui peut expliquer leurs rôles apparemment divers dans le développement embryonnaire, l’obésité, le diabète, la physiologie musculaire et les néoplasmes. De futures études permettront de mieux caractériser les liens entre les gènes à empreinte et les gènes de la matrice extracellulaire.
Principales publications
• Varrault A et al. (2017) Nucleic Acids Res, 45, 10466.
• Al Adhami H et al. (2015) Genome Res, 25, 353
• Varrault A et al. (2006) Dev Cell, 11, 711.
Financements
• 2019-2021 Labex EpiGenMed-Partner
• 2007-2011 ANR 2007-EpiNet-Coordinator
Collaborations
• Paul Pavlidis (Vancouver)
Alumni
• Hala Al Adhami (PhD student, 2008-2012)
• Brendan Evano (PhD student, 2008-2012)


